دکتر گرگ مک کالوم ما را در پشت صحنه آخرین انتشار خود، ‘راهبردهای کاهش نویز در ضبط تایید کروموزوم متاژنومی برای پیوند ژن های مقاومت آنتی بیوتیکی به میزبان های میکروبی می برد‘ منتشر شده در Microbial Genomics.
نام من دکتر گرگ مک کالوم است، من در حال حاضر یک دانشیار تحقیقاتی فوق دکترا در دانشگاه لیورپول، انگلستان هستم. من دکترای خود را در مؤسسه میکروبیولوژی و عفونت، دانشگاه بیرمنگام، انگلستان، در آزمایشگاه پروفسور ویلم ون شایک به پایان رساندم، جایی که در مورد مقاومت آنتی بیوتیکی در میکروبیوتای روده انسان تحقیق کردم.
مقاومت آنتیبیوتیکی، جایی که باکتریها در برابر داروهایی که ما برای درمان عفونت استفاده میکنیم، مقاوم میشوند، یکی از بزرگترین تهدیدهای سلامت جهانی است که بشریت با آن مواجه است. از آنجایی که باکتری های بیشتری در برابر آنتی بیوتیک ها مقاوم می شوند، تعداد افرادی که به دلیل عفونت ها جان خود را از دست می دهند به سرعت در حال افزایش است. جهشهای تصادفی DNA میتوانند به باکتریها اجازه دهند در برابر آنتیبیوتیکها مقاومت کنند. اما مهمتر از آن، ژن های مقاوم به آنتی بیوتیک می توانند بین باکتری ها روی قطعات کوچک DNA به نام پلاسمید پخش شوند. این فرآیند انتقال افقی ژن نامیده می شود و در بسیاری از اکوسیستم ها از جمله میکروبیوتای روده انسان رخ می دهد.
میکروبیوتای روده انسان از صدها گونه باکتری تشکیل شده است که در مجرای روده انسان زندگی می کنند. بیشتر این باکتری ها با میزبان خود رابطه ای مشترک یا متقابل دارند. بنابراین، انسان از میکروبیوتای روده که به هضم غذا کمک می کند و به سیستم ایمنی بدن کمک می کند، سود می برد. با این حال، میکروبیوتای روده همچنین حاوی پاتوژن های فرصت طلب است. این پاتوژنهای فرصتطلب گونههایی هستند که میتوانند بدون ایجاد آسیب در روده یک فرد سالم زندگی کنند، اما در برخی موارد میتوانند از فرصت برای ایجاد عفونت استفاده کنند، زمانی که میزبان دچار نقص ایمنی یا آسیبپذیر شود. در چند دهه اخیر، افزایش قابل توجهی در تعداد عفونتهای ناشی از پاتوژنهای فرصتطلب مقاوم به آنتیبیوتیک که از روده منشا میگیرند، وجود داشته است.
از آنجایی که میکروبیوتای روده در محیطی زندگی میکند که انتقال افقی ژن را تشویق میکند، این فرضیه وجود دارد که پاتوژنهای فرصتطلب ممکن است مقاومت آنتیبیوتیکی را از باکتریهای معمولی کسب کنند. تحقیقات میکروبیوتای روده اغلب از توالی یابی متاژنومیک استفاده می کند، که در آن تمام DNA از یک نمونه پیچیده توالی یابی می شود تا آنچه وجود دارد را پیدا کند. این فناوری نشان داده است که میکروبیوتای روده انسان حاوی بسیاری از ژن های مقاومت آنتی بیوتیکی است که در مجموع به آنها مقاومت روده انسان می گویند. توالی یابی متاژنومیک همچنین اطلاعاتی از گونه های باکتری موجود در نمونه را ارائه می دهد. با این حال، پیوند دادن این دادهها برای مشاهده اینکه کدام باکتری حامل آن ژنهای مقاومت هستند، امری بیاهمیت نیست، بهویژه اگر ژن مقاومت روی پلاسمید جدا از کروموزوم باکتری وجود داشته باشد.
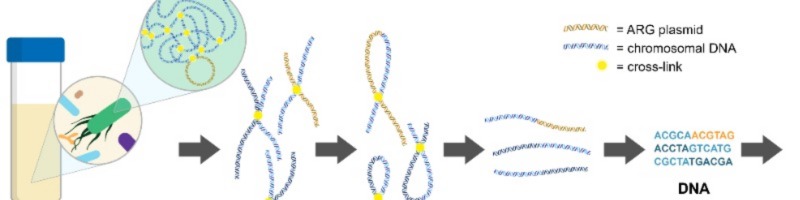
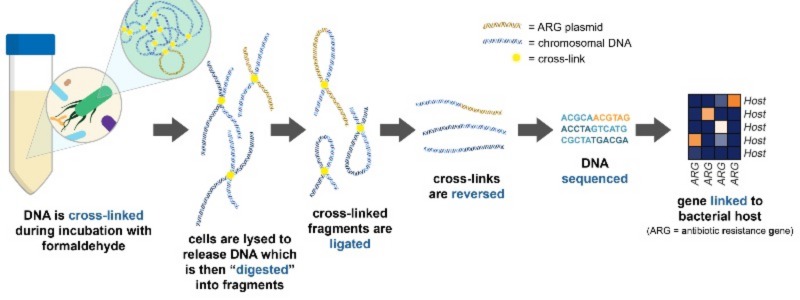
دکترای من بر روی استفاده از تکنیکی به نام جذب ترکیب کروموزوم متاژنومیک (که meta3C یا Hi-C نامیده می شود)، برای پیوند دادن ژن های مقاومت به میزبان خود برای مشاهده میزان حمل و انتقال ژن های مقاومت آنتی بیوتیکی در روده انسان توسط باکتری های مشترک ما متمرکز بود. Meta3C/Hi-C با اتصال فیزیکی DNA در داخل سلولها قبل از تعیین توالی کار میکند و به ما امکان میدهد پیوندهایی بین ژنهای مقاومت آنتیبیوتیکی و قطعات DNA کروموزومی پیدا کنیم که سپس میتوان از آن برای شناسایی گونههای باکتری حامل ژن استفاده کرد.
نحوه عملکرد meta3C/Hi-C. DNA قبل از توالی یابی به صورت فیزیکی به هم مرتبط می شود و به ما این امکان را می دهد که میزبان های باکتریایی توالی های خاصی از DNA را با یافتن این پیوندها در داده ها بررسی کنیم. برگرفته از شکل 1.
کار اخیراً منتشر شده ما بر ایجاد یک گردش کار برای تجزیه و تحلیل دادههای meta3C/Hi-C برای پیوند ژنهای مقاومت به میزبانهای باکتریایی آنها در روده انسان متمرکز بود. در طول تجزیه و تحلیل دادههای meta3C خود بر روی یک نمونه مدفوع انسانی، ما در نتایج به آثاری دست یافتیم که میتواند ژنها را به اشتباه به گونهای نادرست مرتبط کند. در حین توسعه گردش کار آنالیز، ما دادههای سایر مطالعات منتشر شده را که متا3C/Hi-C را بر روی نمونههای میکروبیوتای روده انجام میدادند، دوباره تجزیه و تحلیل کردیم و دریافتیم که این مصنوعات در همه دادههای meta3C/Hi-C وجود دارد. به منظور کاهش تأثیر این مصنوعات، ما مراحلی را برای حذف نویز در داده ها در طول تجزیه و تحلیل، از جمله حذف پیوندها به مناطق تکراری ژنوم، و نیاز به حداقل پنج پیوند منحصر به فرد بین توالی ها برای در نظر گرفتن پیوند “واقعی” ایجاد کردیم. .
با استفاده از این جریان کار تجزیه و تحلیل جدید، ما توانستیم 87 ژن مقاومت آنتی بیوتیکی را به میزبان های باکتریایی آنها در مجموعه داده ها پیوند دهیم. این پیوندها نشان داد که ژنهای کدکننده مقاومت به آنتیبیوتیکهای آمینوگلیکوزید و تتراسایکلین در گونههای معمولی روده انسان گسترده هستند، که نشان میدهد باکتریهای روده یک مخزن مهم برای ژنهای مقاومت هستند.
هدف کار آینده توسعه بیشتر این گردش کار آنالیز برای بهبود طبقهبندی میزبانهای مرتبط است تا میزبانهای باکتریایی بیشتری از ژنهای مقاومت آنتیبیوتیکی آشکار شوند. این ویژگی مقاومت روده انسان همچنان برای یافتن اینکه چگونه پاتوژنهای فرصتطلب در برابر آنتیبیوتیکها مقاوم میشوند، مهم است، بنابراین بهتر میتوانیم بدانیم چگونه از ظهور گونههای مقاوم به دارو که سلامت جهانی آینده را تهدید میکنند، جلوگیری کنیم.